Emissionen aus Schweinemastanlagen
Zusammenfassung Das Landesamt für Natur, Umwelt und Verbraucherschutz Nordrhein-Westfalen (LANUV NRW) und die Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA) untersuchten in einem gemeinsamen Projekt an zehn Schweinemastanlagen, ob und in welchem Umfang antibiotikaresistente Bakterien in der Abluft der Ställe auftreten. Methicillin-resistente Staphylococcus aureus wurden an fünf Ställen nachgewiesen. Trimethoprim/Sulfamethoxazol-resistente Stämme von Enterococcus faecalis waren in zwei Proben detektierbar, allerdings nur in Konzentrationen unterhalb der Bestimmungsgrenze. Trimethoprim/Sulfamethoxazol-resistente E. faecium, Erythromycin-resistente Streptococcus spp. und ESBL-bildende Spezies aus der Familie der Enterobacteriaceae (z. B. Escherichia coli) konnten durch Kultivierung nicht gefunden werden. Des Weiteren wurden Erkenntnisse über die Zusammensetzung der Bakteriengemeinschaften in den Bioaerosolproben gewonnen. Auf der Basis molekularbiologischer Untersuchungen wurde festgestellt, dass Bakterien der Gattungen Clostridium und Lactobacillus dominant in der Abluft der Schweinemastställe auftreten. Außerdem wurden weitere Hinweise über die Eignung kommerziell erhältlicher Selektivnährböden zum quantitativen Nachweis bestimmter Bakterienarten oder -gattungen erhalten.

Massentierhaltung in den Monti Lessinis
Foto: PantherMedia/Christa Eder
1 Einführung
Bakterien sind in der Natur allgegenwärtig, sie kommen natürlicherweise in allen Lebensräumen wie Wasser, Boden und Luft vor und besiedeln auch Mensch und Tier. Besonders hohe Konzentrationen luftgetragener Bakterien treten bei der Haltung von Tieren mit hoher Belegungsdichte auf, wie sie bei der Nutztierhaltung üblich ist. Hier können die Konzentrationen von Bakterien gegenüber den üblichen Werten in der Außenluft um mehrere Größenordnungen erhöht sein. Solche Anlagen geraten zunehmend in den Fokus öffentlicher Diskussionen, vor allem aufgrund ungeklärter Fragen zur Gesundheitsgefährdung von Beschäftigten und Anwohnern, die möglicherweise den emittierten Bakterien ausgesetzt sind. In diesem Zusammenhang kommt derzeit der Frage nach dem Austrag luftgetragener antibiotikaresistenter Bakterien eine besondere Bedeutung zu. Ähnlich wie im Gesundheitswesen, wo die Problematik der Antibiotikaresistenz ein gravierendes und schon länger bekanntes Problem ist, können auch die im Bereich der Tierhaltung verabreichten Medikamente zur Selektion resistenter Bakterien beitragen. Bei einer Infektion des Menschen mit solchen resistenten Bakterien besteht die Gefahr, dass medizinische Behandlungen langwieriger und aufwendiger werden.
Aus nationalen und internationalen wissenschaftlichen Untersuchungen ist z. B. bekannt, dass Methicillin-resistente Staphylococcus aureus (MRSA) in der Nutztierhaltung auftreten können. Hiervon sind insbesondere Schweinemastbetriebe betroffen. Im Rahmen des Verbundprojekts „MRSA-Problematik in der Nutztierhaltung“ des Bundesministeriums für Bildung und Forschung (BMBF) wurde MRSA häufig in Tierbeständen sowie in der Stallluft, aber auch im Außenbereich in der näheren Umgebung von Ställen in Luft- und Bodenproben nachgewiesen. Es konnte weiterhin gezeigt werden, dass Personen mit engem Tierkontakt, wie Landwirte und Beschäftigte, häufig mit MRSA besiedelt sind [1 bis 6].
Innerhalb bestimmter Bakterienarten wird zunehmend die Bildung spezieller Enzyme beobachtet, durch die diese Bakterien Resistenzen gegenüber fast allen Beta-Laktamantibiotika entwickeln. Diese Enzyme werden als Extended-Spektrum-Beta-Laktamasen (ESBL) bezeichnet. Aufgrund des Nachweises von ESBL-bildenden Enterobacteriaceae in der Geflügelstallluft sowie in Bodenproben im Umfeld dieser Ställe wurde ein möglicher Austrag dieser Arten aus den Tierhaltungsanlagen auch im Zusammenhang mit dem Luftweg diskutiert [7; 8]. Kürzlich wurden erstmals auch Ergebnisse einer Studie an Schweinemastställen veröffentlicht, die vermuten lassen, dass ein Austrag von ESBL-bildenden Enterobacteriaceae aus Schweinemastanlagen über Fäkalien, Mäuse und Fliegen, aber auch über die Luft stattfinden könnte [9; 10]. Da an diesen Anlagen bisher noch keine standardisierten Emissionsmessungen durchgeführt wurden, bzw. insgesamt nur sehr wenige Studien existieren, die der Beantwortung dieser Frage nachgegangen sind, gibt es derzeit noch keine konkreten Angaben über die Bedeutung des Luftpfades für eine Verbreitung von ESBL-bildenden Enterobacteriaceae.
In den letzten Jahren hat das Landesamt für Natur, Umwelt und Verbraucherschutz (LANUV) Nordrhein-Westfalen in Zusammenarbeit mit der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA) mehrere Untersuchungsvorhaben zum Emissionsverhalten von Bioaerosolen durchgeführt. Insbesondere wurden repräsentative Messungen an zwangsbelüfteten Hähnchenmastanlagen vorgenommen, um die Emissionen von Mikroorganismen aus quantitativer und qualitativer Sicht näher zu charakterisieren [11 bis 13]. MRSA konnte hierbei nur an einem von acht untersuchten Ställen festgestellt werden, wogegen in allen Ställen Trimethoprim/Sulfamethoxazol-resistente Stämme von Enterococcus faecalis nachgewiesen wurden, allerdings jeweils nur in sehr geringen Konzentrationen unterhalb der Bestimmungsgrenze.
Ziel des hier beschriebenen Projekts war es, zu untersuchen, ob und in welchem Umfang MRSA sowie weitere antibiotikaresistente Bakterien, die für den Menschen gesundheitsrelevant sein können, wie z. B. resistente Enterococcus faecalis und Enterococcus faecium, Enterobacteriaceae und Streptococcus spp. in der Abluft von Schweinemastställen auftreten. Die Untersuchungen sollten letztlich Hinweise auf die Bedeutung des Luftpfads für die Ausbreitung resistenter Bakterien in die Umgebung der betroffenen Anlagen geben. Weiterhin sollten die bisher vorliegenden Erkenntnisse über die Zusammensetzung der Bakteriengemeinschaften verbessert werden, um ggf. weitere Leitparameter für Schweinemastanlagen zu identifizieren. Weiterhin sollten Informationen über die Eignung kommerziell erhältlicher Selektivnährböden zum quantitativen Nachweis der Zielorganismen erhalten werden.
2 Anlagen, Material und Methoden
2.1 Ausgewählte Anlagen
Alle Messungen erfolgten an beheizbaren und zwangsbelüfteten Ställen oder Betriebseinheiten gemäß Nr. 5.4.7.1 der TA Luft (Anlagen zum Halten oder zur Aufzucht von Nutztieren) mit einem Tierbestand von bis zu 1 440 Schweinen. Die Tiere wurden in Einflächenbuchten mit vollperforierten Böden und Flüssigentmistung ohne Einstreu in Klein- oder in Großgruppen gehalten, was der üblichen Haltungstechnik entspricht. Keiner der Mastställe verfügte über eine Abluftreinigungsanlage. Da die Beteiligung der Landwirte auf freiwilliger Basis erfolgte, war die Auswahl der Anlagen nicht zwangsläufig repräsentativ. Es wurde jedoch darauf geachtet, dass nur Anlagen in den Untersuchungsumfang einbezogen wurden, die typisch für die Schweinemast sind.
2.2 Durchführung der Messungen
Alle Messungen erfolgten nach dem Stand der Technik unter Anwendung einschlägiger VDI-Richtlinien im Zeitraum von Oktober 2013 bis Februar 2015. An jedem Stall wurden jeweils an einem Tag vier bis fünf Probenahmen mit einer Dauer von jeweils 30 min gemäß VDI 4257 Blatt 2 [14] durchgeführt. Insgesamt fanden an zehn Messtagen 43 Einzelmessungen statt und es wurden zur Qualitätssicherung 17 Feld- und Laborblindwertproben erzeugt. Die Proben wurden nach Beendigung eines Messtages mit einem Transportunternehmen unter kontrollierten Bedingungen gekühlt zur Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA) nach Berlin transportiert, wo am darauffolgenden Tag mit den mikrobiologischen Aufarbeitungen begonnen wurde.
Zusätzlich wurden an jedem Messtag neben den Abluftrandbedingungen und dem Gesamtabluftvolumenstrom weitere Parameter wie Anzahl, Alter und Masse der Tiere erfasst.
2.3 Analytik
Die vier bis fünf Emissionsproben eines Messtages wurden zunächst zu einer Mischprobe vereint, homogenisiert und in Aliquote aufgeteilt, aus denen die nachfolgenden analytischen Untersuchungen vorgenommen wurden.
2.3.1 Bestimmung der Gesamtzellzahl
Bei allen Proben erfolgte eine Bestimmung der Gesamtzellzahl nach den Vorgaben der Richtlinie VDI 4253 Blatt 4 durch Anfärbung mit dem Fluoreszenzfarbstoff DAPI [15]. Dies dient ebenso wie das Mitführen von Feldblindwertproben der Qualitätssicherung der Messungen, da der Parameter Gesamtzellzahl auch in den vorangegangenen Untersuchungen an Schweinemastanlagen ermittelt wurde und entsprechende Vergleichsdaten vorliegen.
2.3.2 Qualitative Bestimmung antibiotikaresistenter Bakterienarten
Als Zielorganismen oder Zielorganismengruppe wurden A) Staphylococcus aureus, B) Enterococcus faecalis und E. faecium, C) Streptococcus spp. sowie D) Spezies der Familie Enterobacteriaceae (z. B. Escherichia coli) festgelegt, die zum einen im Vorfeld in der Abluft von Schweinemastanlagen detektiert wurden oder im Rahmen von Untersuchungen zu Bioaerosolen im Bereich der Schweinemast als relevante Expositionsparameter festgestellt wurden und im Zusammenhang mit negativen gesundheitlichen Effekten beschrieben sind.
Die Bestimmung antibiotikaresistenter Bakterien erfolgte durch Kultivierung eines Aliquots der Mischprobe auf verschiedenen Selektivnährmedien, die einen Antibiotikazusatz enthielten (siehe Tabelle 1).
Hierfür wurde zunächst eine dezimale Verdünnungsreihe der Mischprobe bis zur Verdünnungsstufe 10-7 angelegt. Dieser Arbeitsschritt ist erforderlich, um für jeden Zielorganismus auswertbare Platten zu erhalten, d. h. es sollten weder zu viele (ca. > 150) noch zu wenige (< 10) Bakterienkolonien vorhanden sein. Aus jeder Verdünnungsstufe wurden pro Nährmedium drei Platten mit jeweils 0,1 ml Bakteriensuspension beimpft und bei 37 °C inkubiert. Um den Anteil der antibiotikaresistenten Bakterien an den kultivierbaren Zielorganismen zu ermitteln, wurden zusätzlich Proben aller Verdünnungsstufen auf Nährmedien ohne Zusatz von Antibiotika ausplattiert. Somit wurden insgesamt pro Mischprobe 192 Nährmediumplatten beimpft. Des Weiteren wurden Feldblindwert- und Laborblindwertproben auf Selektivmedien ohne Antibiotikumzusatz ausplattiert und ebenfalls im Brutschrank bei 37 °C inkubiert.
Ließen sich mit der beschriebenen Methode keine resistenten Bakterien nachweisen, wurden zusätzlich Proben in konzentriertem Flüssignährmedium mit Antibiotikumzusatz vorangereichert und, falls es zum Bakterienwachstum kam, erneut kultiviert. Eine Quantifizierung der Bakterien war dann aber methodisch bedingt nicht mehr möglich.
Aufgrund der Bakterienvielfalt in Umweltproben und der Erkenntnisse aus dem vorangegangenen Projekt zur Ermittlung antibiotikaresistenter Bakterien an Hähnchenmastanlagen [13] war zu erwarten, dass auf den ausgewählten Selektivnährmedien nicht nur Zielorganismen kultiviert werden können, sondern die Wachstumsbedingungen auch für andere Bakterien geeignet sind. Es ist deshalb erforderlich, die aufgetretenen Kolonien weitergehend zu untersuchen und eine eindeutige Identifizierung zu Bakteriengattung und/oder -art vorzunehmen.
Hierfür wurden pro Selektivnährmedium mit Antibiotikazusatz mindestens drei morphotypische Bakterienkolonien ausgewählt. Ein Teil des Zellmaterials wurde jeweils auf Glasobjektträger verbracht, mikroskopisch beurteilt und fotografiert. Um die Zellen zu vereinzeln, wurden weitere Nährmedienplatten durch fraktionierten Ausstrich der ausgewählten Kolonien beimpft und wiederum bebrütet. Die durch diese Technik gewonnenen Einzelkolonien wurden abermals mikroskopiert, fotografiert und erneut übertragen. Diese Vorgehensweise wurde so lange, mindestens jedoch fünfmal, wiederholt, bis bei der mikroskopischen Analyse ein einheitlicher Zelltyp erkennbar war. Die so erhaltenen Bakterienisolate wurden anschließend weitergehend charakterisiert und identifiziert. Die Identifizierung der Reinkulturen erfolgte dabei anhand von 16S-rRNS-Genanalysen durch Vergleich mit in Datenbanken hinterlegten Gensequenzen. Hierfür wurde die genomische DNS aus einem Teil der Reinkulturen extrahiert und anschließend das Gen der 16S-rRNS über eine Polymerase-Kettenreaktion (PCR) vervielfältigt. Durch eine Sequenzierung der PCR-Produkte wurde die Nukleinsäuresequenz des Gens ermittelt. Dabei wurden die von Martin et al. beschriebenen Methoden zugrunde gelegt [16]. Die beiden pro Bakterium erhaltenen Teilsequenzen wurden zunächst vereinigt und anschließend mit den Nukleinsäuresequenzen von Typstämmen bekannter Bakterienarten, die in öffentlichen Sequenzdatenbanken zur Verfügung stehen (in silico), verglichen (Ribosomal Database [17] oder GenBank [18]).
Ergänzend zur 16S-rRNS-Genanalyse wurde ein für die klinische Diagnostik verwendetes System (VITEK®2TM Compact System, Fa. bioMérieux) zur Identifizierung eingesetzt, das die Verwertung unterschiedlicher Substrate und die Präsenz von Enzymen als Datengrundlage nutzt, um die Reaktionsmuster von Isolaten mit denen von klinisch relevanten Arten zu vergleichen. Alle gewonnenen Zielorganismen wurden mit dem VITEK®2TM-System gegenüber einer Vielzahl unterschiedlicher Antibiotika auf ihre Resistenzeigenschaften getestet.
2.3.3 Zusammensetzung der Bakteriengemeinschaft in der Emissionsprobe auf Gattungsebene und Ähnlichkeiten zu Bakterienspezies
Zur Untersuchung der Bakterienvielfalt in der Abluft von Schweinemastanlagen wurde bakterielle DNS aus einem Aliquot der Mischproben extrahiert und mithilfe der Polymerase-Kettenreaktion die darin enthaltenen 16S-rRNS-Gene vervielfältigt. Die PCR erfolgte dabei mit universellen Primern [19] unter den von Martin et al. [12] beschriebenen Bedingungen. Für jede Mischprobe wurden für den Fall eines positiven PCR-Produktnachweises die 16S-rRNS-Gene durch das Anlegen einer Klonbibliothek vereinzelt. Aus dieser Genbibliothek wurden 300 16S-rRNS-Gene zufällig ausgewählt und sequenziert. Die Zuordnung bzw. Identifizierung der DNS-Sequenzen erfolgte über den In-silico-Vergleich mit 16S-rRNS-Gensequenzen bekannter bakterieller Typstämme.
3 Ergebnisse und Diskussion
In Tabelle 2 ist die Belegung der Mastställe bzw. der untersuchten Betriebseinheiten mit Schweinen zum Zeitpunkt der Emissionsmessungen aufgeführt.
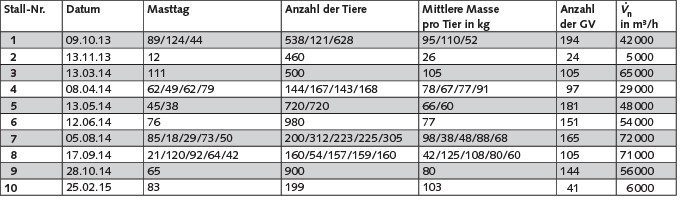
Tabelle 2. Daten zur Stallbelegung zum Zeitpunkt der Emissionsmessungen und ermittelte Gesamtabluftvolumenströme. GV = Großvieheinheiten
Die Tabelle zeigt für jeden Messtag den Masttag, die entsprechende Anzahl der Tiere, die mittlere Einzeltiermasse und die daraus resultierende Anzahl der Großvieheinheiten im Maststall bzw. in der Betriebseinheit. Die Großvieheinheiten (GV) berechnen sich aus der anzahlgewichteten Gesamttiermasse und der Gesamttierzahl. Eine GV dient als Umrechnungsschlüssel zum Vergleich verschiedener Nutztiere auf der Basis ihres Lebendgewichts und entspricht dabei 500 kg Tierlebendgewicht. Zusätzlich gibt die Tabelle für jeden Stall die Gesamtabluftvolumenströme (in Norm, trocken) wieder, gemittelt über die Dauer der gesammelten Einzelproben. Die Werte schwanken etwa um eine Größenordnung; sie sind von der Anzahl der im jeweiligen Stall befindlichen Tiermasse sowie von der Außentemperatur abhängig und als plausibel einzustufen.
Die Anzahlkonzentrationen der Gesamtzellen in der Abluft variierten nur geringfügig zwischen 2 107 und 6 107 Zellen/m³. Diese Werte sind vergleichbar mit Ergebnissen, die in anderen Studien mittels PCR-Verfahren oder bei fluoreszenzmikroskopischen Analysen in der Stallluft ermittelt wurden [20 bis 24].
Die spezifischen Emissionen bezogen auf Großvieheinheiten variierten zwischen 1 106 und 7 106 Zellen/(GVs). Vergleichbare Werte wurden auch bei vorangegangenen Untersuchungen an Schweinemastanlagen festgestellt [25], sodass die Ergebnisse als plausibel zu bewerten sind.
3.1 Resistente Staphylococcus aureus
Nach Kultivierung auf dem verwendeten Selektivnährmedium für Staphylococcus aureus mit und ohne Antibiotikumzusatz wurden zunächst bei Proben aus allen zehn Ställen „verdächtige“ Kolonien festgestellt, bei denen es sich aufgrund ihrer charakteristischen Form und Farbe um den Zielorganismus handeln könnte. Diese sollen gemäß Herstellerangaben auf dem Nährböden als pink- bis mauvefarbige Kolonien erscheinen. Das Bild zeigt die aus den Zählergebnissen, den Verdünnungsfaktoren, den Impingervolumina und den jeweils abgesaugten Teilvolumina der Abluft berechneten Konzentrationen verdächtiger Staphylococcus aureus und verdächtiger MRSA.
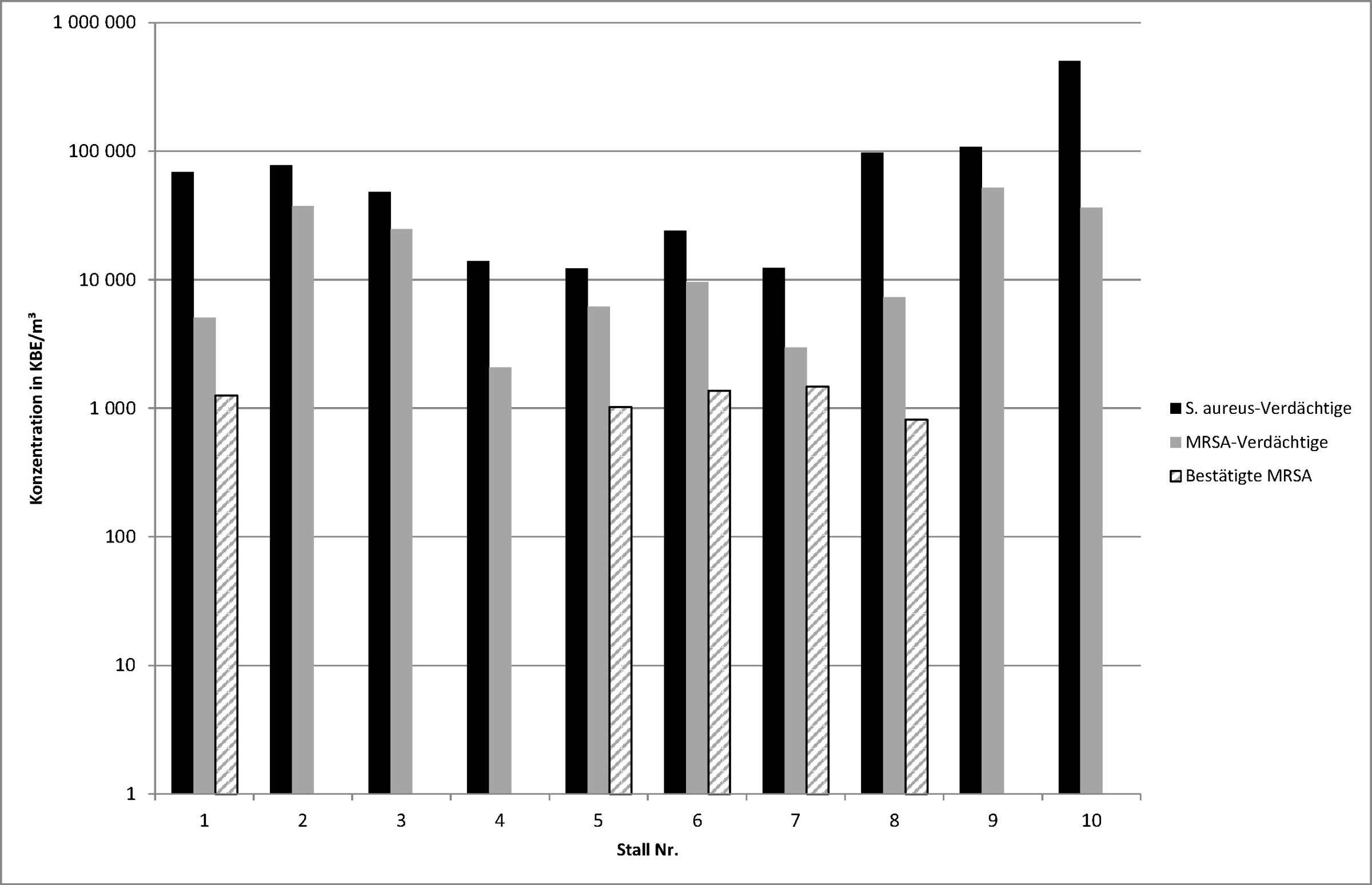
Konzentrationen in KBE/m3 verdächtiger Staphylococcus aureus, verdächtiger MRSA sowie bestätigter MRSA in den zehn untersuchten Ställen.
Foto: LaNuV NRW / BAuA
Die weitergehenden biochemischen (VITEK® 2TM Compact System) und molekularbiologischen Untersuchungen (16S-rRNS-Genanalysen) zur Identifizierung der MRSA-verdächtigen Kolonien ergaben, dass MRSA-Stämme in fünf der zehn Mischproben tatsächlich vorhanden waren. Der Anteil der MRSA-emittierenden Ställe an der Gesamtzahl der untersuchten Ställe ist damit bei der Schweinemast deutlich höher als bei der Hähnchenmast [13]. Die hier erzielten Ergebnisse bestätigen somit die bisherigen Untersuchungen zur Prävalenz von MRSA in Schweinemastställen, die zwischen 30 bis über 70 % der untersuchten Schweineherden als MRSA-positiv bezifferten [26 bis 30].
Die Ergebnisse verdeutlichen auch, dass in Bioaerosolproben aus der Abluft von Schweinemastställen eine Vielzahl von Bakterien vorhanden sein kann, die sich auf dem verwendeten Selektivnährboden CHROMagarTM MRSA kultivieren lassen und deren koloniemorphologisches Erscheinungsbild eine einfache Differenzierung vom Zielorganismus nicht ohne Weiteres erlaubt. Eine nachfolgende Identifizierung der aufgetretenen Kolonien ist daher unerlässlich, um belastbare Aussagen zum qualitativen und quantitativen Auftreten von MRSA treffen zu können.
Die Abschätzung der in den Proben vorliegenden MRSA-Konzentrationen erfolgte über das Verhältnis von bestätigten zu verdächtigen MRSA-Isolaten. Da diese auf den auswertbaren Platten jeweils nur in geringer Anzahl vorlagen, ist die quantitative Bestimmung der Konzentration des Zielorganismus MRSA auf der Grundlage der Zählergebnisse der auf Selektivnährböden gewachsenen Bakterienkolonien mit einer nicht näher bezifferbaren hohen statistischen Unsicherheit behaftet. Als Schätzwert kann für die fünf Ställe jeweils eine Konzentration von etwa 10³ KBE MRSA-Stämmen pro m³ Abluft angenommen werden. Diese Werte befinden sich innerhalb der Konzentrationsspanne, die von anderen Autoren bei Untersuchungen innerhalb der Schweinemastställe beobachtet wurde [3; 4; 29].
Die bestätigten MRSA-Isolate wurden auch auf Resistenzbildung gegenüber weiteren Antibiotikaklassen untersucht. Hierbei wurden Resistenzen gegenüber Enrofloxacin (Chinolon), Imipenem (Carbapenem) und Tetracyclin festgestellt, sodass Resistenzmechanismen auch gegenüber anderen Antibiotikaklassen als β-Lactamantibiotika vorhanden sind.
Bei den anderen Reinkulturen, die vom Nährmedium CHROMagarTM MRSA isoliert wurden, handelte es sich nicht um MRSA. Die insgesamt untersuchten 65 Isolate waren auf der Grundlage der Analyse der 16S-rRNS-Gene folgenden Bakterienarten am ähnlichsten:
- Aerococcus viridans in acht Mischproben,
- Rothia nasimurium in fünf Mischproben.
- Staphylococcus pasteuri in vier Mischproben,
- Staphylococcus cohnii in drei Mischproben,
- Aerococcus suis, Bacillus licheniformis, Bacillus galactosidilyticus, Brevibacterium avium in jeweils zwei Mischproben,
- Aerococcus urinaeequi, Corynebacterium mycetoides, Luteococcus sediminum, Globicatella sulfidifaciens, Staphylococcus haemolyticus, Staphylococcus jettensis, Staphylococcus epidermidis, Kurthia huakuii, Microbacterium ginsengiterrae, Kocuria palustris, Rothia amarae, Brevibacterium epidermidis und Jeotgalicoccus halotolerans in jeweils einer Mischprobe.
Einige der isolierten Organismen besitzen auf der Basis ihrer 16S-rRNS-Sequenzen die höchste Ähnlichkeit zu den Bakterien Aerococcus viridans, Aerococcus suis, Brevibacterium avium, Globicatella sulfidifaciens, Staphylococcus pasteuri, Staphylococcus haemolyticus, Staphylococcus epidermidis und Corynebacterium mycetoides, die gemäß der Technischen Regel für Biologische Arbeitsstoffe (TRBA) 466 [31] in Risikogruppe 2 eingestuft sind.
Auf der Grundlage der Untersuchungen kann keine Aussage darüber getroffen werden, ob die nicht als MRSA identifizierten Spezies dasselbe Gen tragen, das in Staphylococcus aureus für die Methicillin-Resistenz verantwortlich ist, oder ob andere Resistenzmechanismen vorhanden sind, die ein Wachstum auf dem antibiotikahaltigen Selektivnährboden ermöglichen.
3.2 Enterococcus faecalis und Enterococcus faecium
Die Herstellung von Reinkulturen zum Nachweis der Zielorganismen Enterococcus faecalis und Enterococcus faecium erfolgte auf einem CATC-Agar unter Zugabe des Antibiotikagemischs Trimethoprim/Sulfamethoxazol. Bei keiner Probe konnten verdächtige Bakterienstämme durch direkte Kultivierung gewonnen werden. In zwei der zehn untersuchten Proben wurde aus der Anreicherungskultur Enterococcus faecalis nachgewiesen. Antibiotikaresistente Enterococcus-faecium-Stämme waren in keiner der Proben identifizierbar.
Alle drei Enterococcus-faecalis-Isolate zeigten Resistenzen gegenüber Trimethoprim/Sulfamethoxazol. Dieses Ergebnis ist plausibel, da die Kombination dieser Antibiotika zur Selektion dem Kulturmedium zugesetzt wurde. Die Isolate zeigten außerdem Resistenzen gegenüber den Antibiotika Enrofloxacin, Erythromycin und Clindamycin. Resistenzen gegenüber dem Reserveantibiotikum Vancomycin konnten hingegen nicht festgestellt werden.
Auch der verwendete CATC-Agar ist somit nicht vollständig spezies- und gattungsselektiv. Die übrigen 15 gewonnenen Isolate waren auf der Basis des 16S-rRNS-Gens am ähnlichsten zu
- Staphylococcus cohnii in vier Mischproben,
- Enterococcus hirae, Enterococcus durans, Enterococcus avium, Aerococcus viridans, Staphylococcus nepalensis, Staphylococcus saprophyticus in jeweils einer Mischprobe.
Bis auf Staphylococcus cohnii handelt es sich bei allen isolierten Nichtzielbakterienarten um solche, deren 16S-rRNS-Gen am ähnlichsten zu Vertretern von Mikroorganismen der Risikogruppe 2 ist. Auch hier bleibt die Frage offen, welche Resistenzeigenschaften diese Isolate gegenüber den eingesetzten Antibiotika besitzen.
Eine geringe Spezifität beim kulturabhängigen Nachweis von Vertretern der Gattung Enterococcus wurde auch von Chapin et al. [32] auf einem modifizierten m-Enterococcus- Agar beobachtet. Unter den 137 von dieser Arbeitsgruppe als Enterococcus verdächtig isolierten Bakterienspezies, handelte es sich bei nur 34 % um Vertreter der Gattung Enterococcus; 32 % der Isolate wurden als Koagulase-negative Vertreter der Gattung Staphylococcus identifiziert.
3.3 Streptococcus spp.
Aus allen Emissionsproben konnten bakterielle Reinkulturen gewonnen werden, die auf dem für Streptococcus selektiven COBA-Nährmedium ohne und mit Antibiotikumzusatz wuchsen. Die Konzentration an Bakterien, die auf dem antibiotikumhaltigen COBA-Nährmedium wuchsen, variierte zwischen den Proben von 3 ⋅ 104 bis 5 ⋅ 105 KBE pro m³ Abluft. Die nachfolgenden Identifizierungen von insgesamt 71 Isolaten auf der Basis des 16S-rRNS-Gens haben jedoch gezeigt, dass keines der Isolate der Gattung Streptococcus zuzuordnen war, sondern die Isolate folgenden Bakterienspezies am ähnlichsten waren:
- Aerococcus viridans in neun Mischproben,
- Aerococcus urinaeequi in sieben Mischproben,
- Staphylococcus cohnii in fünf Mischproben,
- Luteococcus sediminum und Rothia nasimurium in jeweils vier Mischproben,
- Corynebacterium freneyi in drei Mischproben,
- Brevibacterium luteolum, Corynebacterium confusum, Corynebacterium xerosis, Jeotgalicoccus pinnipedialis, Staphylococcus saprophyticus und Rothia endophytica in jeweils zwei Mischproben
- Corynebacterium frankenforstense, Corynebacterium stationis, Corynebacterium humireducens, Kocuria atrinae, Kocuria carniphila, Lactococcus formosensis, Staphylococcus haemolyticus, Staphylococcus jettensis und Rothia amarae in jeweils einer Mischprobe.
Ob die identifizierten Spezies die gleichen Resistenzeigenschaften tragen, die bei den Spezies der Gattung Streptococcus für die Erythromycin-Resistenz verantwortlich sind, oder ob andere Resistenzmechanismen vorhanden sind, kann auf der Grundlage der vorliegenden Untersuchungen nicht beantwortet werden. Unter den isolierten Organismen befinden sich auch solche, die auf der Basis ihrer 16S- rRNS-Sequenzen die höchste Ähnlichkeit zu den Bakterien Aerococcus viridans, Staphylococcus haemolyticus, Staphylococcus saprophyticus, Corynebacterium confusum und Corynebacterium freneyi aufweisen, die alle Vertreter der Risikogruppe 2 darstellen.
3.4 Spezies der Familie Enterobacteriaceae (z.B. Escherichia coli)
Auf dem verwendeten Nährmedium mit und ohne Zusatz von Cefotaxim konnte bei keiner der Bioaerosolproben ein Bakterienwachstum verzeichnet werden. In diesem Zusammenhang bestätigen sich frühere Beobachtungen, die zeigen, dass die gramnegativen Enterobacteriaceae als typische Darmbakterien in der Luft schnell abzusterben scheinen.
3.5 Zusammensetzung der Bakteriengemeinschaft in den Emissionsproben
Die bisher durchgeführten molekularbiologischen Untersuchungen in sieben der zehn Mischproben zur Zusammensetzung der emittierten Bakterien zeigten, dass in der Abluft der Schweineställe eine sehr große Vielfalt unterschiedlicher Bakterien vorliegt. Bisher konnten 16S-rRNS- Gensequenzen beobachtet werden, die mehr als 45 unterschiedlichen Bakteriengattungen zuzuordnen sind. Dabei dominierten die Sequenzen mit höchster Ähnlichkeit zu den Bakteriengattungen Clostridium, Lactobacillus, Terrisporobacter, Aerococcus, Turicibacter, Romboutsia, Prevotella, Streptococcus, deren Sequenzen in mindestens fünf der ausgewerteten sieben Proben mit einem durchschnittlichen Anteil von > 1 % auftraten (Tabelle 3).
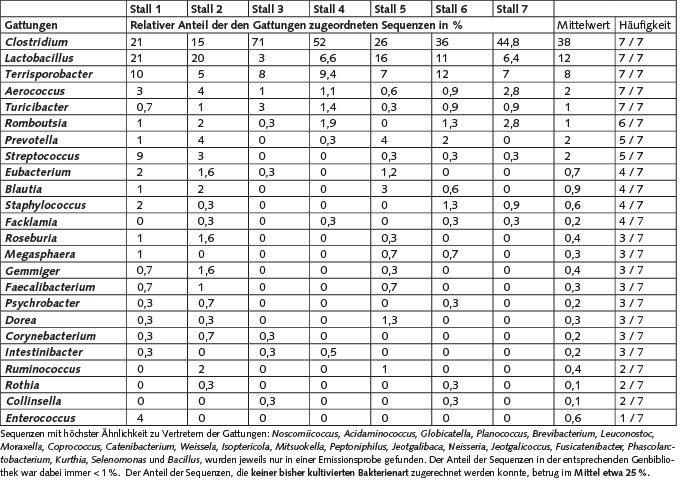
Tabelle 3. Zuordnung der detektieren 16S-rRNA-Gensequenzen auf Gattungsebene und relativer Anteil in % der Sequenzen, der diesen Gattungen zugeordnet wurde. Gattungen, denen Sequenzen nur aus einer Probe zugeordnet werden konnten und deren Anteil in der entsprechenden Genbibliothek < 1% war, sind unterhalb der Tabelle aufgelistet.
Bei der Bewertung dieser Ergebnisse ist zu berücksichtigen, dass die genannten Anteile nicht zwingend das Verhältnis in den Originalproben widerspiegeln, da die hier verwendeten molekularbiologischen Methoden ebenfalls Limitierungen unterliegen können, die bereits von Martin et al. [12] ausführlich diskutiert wurden. So ist bekannt, dass a) das 16S-rRNS-Gen in unterschiedlicher Anzahl in verschiedenen Bakterien vorliegen kann, b) nicht aus allen Bakterienarten die Extraktion der DNS in gleichem Maße gelingt und c) es mit der PCR auch zu selektiven Vervielfältigungen bestimmter Gene kommen kann.
Diese erfassten Gattungen decken sich weitgehend mit Untersuchungen zur Bakterienzusammensetzung, die bereits in Studien von anderen Autoren zur Analyse der Schweinestallluft, der Darmflora von Schweinen und der Bakteriengemeinschaft in Schweinegülle beobachtet wurden [33 bis 35]. Insgesamt lässt sich daraus schlussfolgern, dass sich die Zusammensetzungen der Bakteriengemeinschaften in der Abluft von Schweine- und Hähnchenmastställen deutlich voneinander unterscheiden. Viele Bakterienarten, deren 16S-rRNS-Gensequenzen denen am ähnlichsten sind, die in den Bioaerosolproben detektiert wurden, besitzen beispielsweise einen strikt anaeroben Stoffwechsel. Vertreter der Gattung Clostridium sind außerdem in der Lage, Endosporen zu bilden. Es ist davon auszugehen, dass die Sporen dieser Bakterien aufgrund ihrer Widerstandsfähigkeit den Transport über die Luft und die Probenahme gut überstehen. Daher gilt es, zukünftig zu überlegen und zu untersuchen, ob bestimmte Vertreter der Gattungen Clostridium oder Lactobacillus als Leitparameter genutzt werden können, wenn der Einfluss von Bioaerosolemissionen aus Schweinemastanlagen auf die Immissionsbelastung untersucht werden soll.
Innerhalb der Genbibliotheken lag der Anteil der Sequenzen mit höchster Ähnlichkeit zur Gattung Staphylococcus im Durchschnitt bei etwa 0,6 %. Dieser Wert ist gut vergleichbar mit den Ergebnissen von Masclaux et al. [3] in Bioaerosolproben aus Schweineställen. Hier betrug der mit realtime PCR ermittelte Anteil der Gene aus der Gattung Staphylococcus etwa 0,5 % an der Anzahl aller bakteriellen Gene. Auch die Untersuchungen von Friese et al. [29] zeigten, dass die kultivierbaren Staphylokokken in Bioaerosolen aus Schweineställen mit etwa 5 bis 8 % nur einen geringen Teil der kultivierbaren Bakterien ausmachen. Anders als bei den Emissionen aus Hähnchenmastanlagen, bei denen in Abhängigkeit vom Masttag Anteile von bis über 70 % detektiert wurden [12], gehören Vertreter der Gattung Staphylococcus damit nicht zu den dominanten Vertretern in den Bioaerosolproben der Schweinemast, auch wenn sie regelmäßig von den Kulturmedien isoliert wurden.
Zusammenfassend lässt sich feststellen, dass viele der in der Abluft vorhandenen Bakterien zu solchen gehören, die ausschließlich unter striktem Sauerstoffentzug wachsen können und somit spezielle Anforderungen an ihre Kultivierung stellen. Des Weiteren konnten viele der detektierten 16S-rRNS-Gensequenzen keiner bekannten Bakterienart oder Gattung zugeordnet werden.
4 Resümee
Über die Kultivierung und Identifizierung gewonnener Reinkulturen konnten in der Abluft von fünf der zehn untersuchten Schweinemastställe MRSA-Stämme nachgewiesen werden. Damit decken sich die Ergebnisse mit den bisher vorliegenden Aussagen über ein häufiges Auftreten von MRSA in der Stallluft, im Stallstaub und an untersuchten Tieren. Aufgrund der Anwesenheit weiterer morphotypischer Bakterien auf dem verwendeten Selektivnährmedium CHROMagarTM MRSA ist eine Quantifizierung von MRSA in diesen Proben mit hoher Unsicherheit behaftet. Als grobe Abschätzung der Emissionskonzentration kann ein Wert von 10³ KBE/m³ angegeben werden.
In der Abluft zweier Ställe, allerdings nur aus Anreicherungskulturen, wurden gegenüber Trimethoprim/Sulfamethoxazol resistente Enterococcus-faecalis-Stämme nachgewiesen, die meist auch gegenüber Tetracyclin und Clindamycin resistent waren. Keines dieser Isolate zeigte Resistenzen gegenüber dem Reserveantibiotikum Vancomycin. Trimethoprim/Sulfamethoxazol-resistente Enterococcus-faecium-Stämme, Erythromycin-resistente Streptococcus spp. und ESBL-bildende Enterobacteriaceae konnten in den Bioaerosolproben über den kultivierungsabhängigen Ansatz im Rahmen des Projekts nicht detektiert werden. Es ist somit wenig wahrscheinlich, dass der Luftpfad für die Ausbreitung dieser Bakterien aus quantitativer Sicht eine Rolle spielt.
Inwiefern die Emissionen anderer Bakterienarten der Risikogruppe 2 eine Bedeutung für die Bewertung der Bioaerosolemissionen haben, sollte an anderer Stelle untersucht bzw. geklärt werden. Die molekularbiologischen Untersuchungen bestätigten insgesamt, dass es sich um sehr komplexe Gemische unterschiedlicher Bakterienspezies handelt. Über die Eigenschaften eines Großteils der in der Abluft von Schweinemastanlagen auftretenden Bakterien kann bislang keine Aussage getroffen werden, da ihre Detektion bisher nur auf der Basis molekularbiologischer Methoden gelungen ist. Bei diesen Untersuchungen handelt es sich um einen ersten Ansatz zur Charakterisierung der Bakteriengemeinschaft in der Stallabluft von Schweinemastanlagen. Insgesamt wird aus den Ergebnissen deutlich, dass noch weiterer Untersuchungsbedarf besteht.
Die vorgestellten Untersuchungen stützen auch die bisherigen Erkenntnisse, dass Nährböden nur eingeschränkt für eine quantitative Analyse von Bioaerosolproben mit komplexer Mikroorganismen-Zusammensetzung, wie sie bei Tierhaltungsanlagen auftritt, geeignet sind. Um sicherzustellen, dass es sich bei den Bakterienkolonien tatsächlich um die Zielorganismen handelt, sollte immer eine Isolierung und fachgerechte Identifizierung der Kolonien erfolgen. Das zukünftige Ziel der Normungsarbeit wird es sein, Konventionen für Analysebedingungen festzulegen und in diesem Zusammenhang auch die Messunsicherheiten für die Quantifizierung von Leitparametern zu ermitteln und zu beschreiben.
Danksagung
Wir bedanken uns bei A. Langner, T. Benning, C. Berus, C. Buchner, T. Gras und F. Kotzian für die engagierte Durchführung der Emissionsmessungen. Ferner danken wir herzlichst dem Westfälisch-Lippischen Landwirtschaftsverband e. V. für die Unterstützung bei der Vermittlung geeigneter Anlagen und den beteiligten Landwirten für ihre Bereitschaft, am Untersuchungsprojekt teilzunehmen.
Literatur
- Armand-Levevre, L.; Ruimy, R.; Andremont, A.: Clonal comparison of Staphylococcus aureus isolates from healthy pig farmers, human controls, and pigs. Emerg. Infect. Dis. 11 (2005), S. 711-714.
- de Neeling, A. J.; van den Broek, M. J.; Spalburg, E. C. et al.: High prevalence of methicillin resistant Staphylococcus aureus in pigs. Vet. Microbiol. 122 (2007), S. 366-372.
- Masclaux, F. G.; Sakwinska, O.; Charrière, N.; Semaani, E.; Oppliger, A.: Concentration of airborne staphylococcus (MRSA and MSSA), total bacteria, and endotoxins in pig farms. Ann. Occup. Hyg. 57 (2013) Nr. 5, S. 550-557.
- Schulz, J.; Friese, A.; Klees, S.; Tenhagen, B.-A.; Fetsch, A.; Rösler, U.; Hartung, J.: Longitudinal study of the contamination of air and of soil surfaces in the vicinity of pig barns by livestock-associated Methicillin-resistant Staphylococcus aureus. Appl. Environ. Microbiol. 78 (2012) Nr. 16, S. 5666-5671.
- Denis, O.; Suetens, C.; Hallin, M. et al.: Methicillin-resistant Staphylococcus aureus ST398 in swine farm personnel, Belgium. Emerg. Infect. Dis. 15 (2009) Nr. 7, S. 1098-1101.
- Bisdorff, B.; Scholholter, J. L.; Claussen, K.; Pulz, M.; Nowak, D.; Radon, K.: MRSA-ST 398 in livestock farmers and neighbouring residents in rural area in Germany. Epidemiol. Infect. 140 (2012), S. 1800-1808.
- Li, S.; Zhao, M.; Li, Y.; Zhang, L.; Zhang, X; Miao, Z.: Detection and source identification of airborne extended-spectrum beta-lactamase-producing Escherichia coli isolates in a chicken house. Aerobiologia 29 (2013), S. 315-319.
- Laube, H.; Friese, A.; von Salviati, C.; Guerra, B.; Rösler, U.: Transmission of ESBL/AmpC-producing Escherichia coli from broiler chicken farms to surrounding areas. Vet. Microbiol. 172 (2014), S. 519-527.
- von Salviati, C.; Laube, H.; Guerra, B.; Roesler, U.; Friese, A.: Emission of ESBL/AmpC-producing Escherichia coli from pig fattening farms to surrounding areas. Vet. Microbiol.175 (2015), S. 77-84.
- Gao, L.; Tan, Y.; Zhang, X.; Hu, J.; Miao, Z.; Wie, L.;Tongjie C.: Emissions of Escherichia coli carrying extended-spectrum -lactamase resistance from pig farms to the surrounding environment. Int. J. Environ. Res. Publ. Health 12 (2015) Nr. 4, S. 4203-4213.
- Gärtner, A.; Gessner, A.; Martin, E.; Jäckel, U.: Emissionsmessungen von Mikroorganismen aus Hähnchenmastanlagen – Aktuelle Messergebnisse und vergleichende Untersuchung von drei verschiedenen Ställen. Gefahrstoffe – Reinhalt. Luft 71 (2011) Nr. 9, S. 362-366.
- Martin, E.; Gessner, A.; Gärtner, A.; Jäckel, U.: Molekularbiologische Charakterisierung luftgetragener Bakterien in Emissionsproben aus Hähnchenmastanlagen. Gefahrstoffe – Reinhalt. Luft 72 (2012) Nr. 3, S. 99-103.
- Gärtner, A.; Gessner, A.; Martin, E.; Schneider, D.; Jäckel, U.: Emissionen aus der Hähnchenmast – Untersuchung zur Zusammensetzung der Bakteriengemeinschaft und Antibiotikaresistenz – Teil 2: Ergebnisse. Gefahrstoffe – Reinhalt. Luft 74 (2014) Nr. 9, S. 377-383.
- VDI 4257 Blatt 2: Bioaerosole und biologische Agenzien; Messen von Emissionen; Probenahme von Bioaerosolen und Abscheidung in Flüssigkeiten. Berlin: Beuth 2011.
- VDI 4253 Blatt 4: Bioaerosole und biologische Agenzien – Bestimmung der Gesamtzellzahl mittels Fluoreszenzanalyse nach Anfärbung mit DAPI. Berlin: Beuth 2013.
- Martin, E.; Kämpfer, P.; Jäckel, U.: Erfassung der bakteriellen Diversität in der Innenraumluft. Gefahrstoffe – Reinhalt. Luft 69 (2009) Nr. 3, S. 97-101.
- Cole, J. R.; Wang, Q.; Fish, J. A.; Chai, B.; McGarrell, D. M.; Sun, Y.; Brown, C. T.; Porras-Alfaro, A.; Kuske, C. R.; Tiedje, J. M.: Ribosomal database project: data and tools for high throughput rRNA analysis. Nucl. Acids Res. 42 (2014) Nr. D1, S. D633-D642.
- Benson, D. A.; Cavanaugh, M.; Clark, K.; Karsch-Mizrachi, I.; Lipman, D. J.; Ostell, J.; Sayers, E. W.: Genebank. Nucl. Acids Res. 41 (2013) Nr. D1, S. D36-D42.
- Weisburg, W. G.; Barns, S. M.; Pelletier, P. A.; Lane, D. J.: 16S ribosomal DNA amplification for phylogenetic study. J. Bacteriol. 173 (1991) Nr. 2, S. 697-703.
- Létourneau, V.; Nehmé, B.; Mériaux, A.; Massé, D.; Duchaine, C.: Impact of production systems on swine confinement buildings bioaerosols. J. Occup. Environ. Hyg. 7 (2010) Nr. 2, S. 94-102.
- Verreault, D.; Létourneau, V.; Gendron, L.; Massé, D.; Gagnon, C. A.; Duchaine, C.: Airborne porcine circovirus in Canadian swine confinement buildings. Vet. Microbiol. 14 (2010), S. 224-230.
- Kumari, P.; Choi, H. L.: Seasonal variability in airborne biotic contaminants in swine confinement buildings. PLOS ONE (2014). DOI: 10.1371/journal.pone.0112897
- Nehmé, B.; Gilbert, Y.; Létourneau, V., Forster, R. J., Veillette, M.; Villemur, R.; Duchaine, C.: Culture-independent characterization of archaeal biodiversity in swine confinement building bioaerosols. Appl. Environ. Microbiol. 75 (2009) Nr. 17, S. 5445-5450.
- Bonifait, L.; Veillette, M.; Létourneau, V.; Grenier, D.; Duchaine, C.: Detection of Streptococcus suis in bioaerosols of swine confinement buildings. Appl. Environ. Microbiol. 80 (2014) Nr. 11, S. 3296-3304.
- Gärtner, A.; Gessner, A.; Knust, S.: Ermittlung der Emissionen von Mikroorganismen aus Schweinemastanlagen. Gefahrstoffe – Reinhalt. Luft 74 (2014) Nr. 11/12, S. 505-510.
- Köck, R.; Harlizius, J.; Bressan, N.; Laerberg, R.; Wieler, L. H.; Witte, W.; Deurenberg, R. H.; Voss, A.; Becker, K.; Friedrich, A. W.: Prevalence and molecular characteristics of methicillin-resistant Staphylococcus aureus (MRSA) among pigs on German farms and import of livestock-related MRSA into hospitals. Eur. J. Clin. Microbiol. Infect. Dis. 28 (2009), S. 1375-1382.
- Broens, E. M.; Graat, E. A. M.; van der Wolf, P. J.; van de Giessen, A. W.; de Jong, M. C. M.: Prevalence and risk factor analysis of livestock associated MRSA-positive pig herds in The Netherlands. Prev. Vet. Med. 102 (2011), S. 41-49.
- Alt, K.; Fetsch, A.; Schroeter, A.; Guerra, B.; Hammerl, J. A.; Hertwig, S.; Senkov, N.; Geinets, A.; Mueller-Graf, C.; Braeunig, J.; Kaesbohrer, A.; Appel, B.; Hensel, A.; Tenhagen, B.-A.: Factors associated with the occurrence of MRSA CC398 in herds of fattening pigs in Germany. BMC Vet. Res. 7 (2011), S. 69.
- Friese, A.; Schulz, J.; Hoehle, L.; Fetsch, A.; Tenhagen, B.-A.; Hartung, J., Roesler, U.: Occurrence of MRSA in air and housing environment of pig barns. Vet. Microbiol. 158 (2012) S. 129-135.
- van de Vijver, L. P. L.; Tulinski, P.; Bondt, N.; Mevius, D.; Verwer, C.: Prevalence and molecular characteristics of methicillin-resistant Staphylococcus aureus (MRSA) in organic pig herds in The Netherlands. Zoonoses Publ. Health 61 (2014), S. 338-345.
- Technische Regeln für Biologische Arbeitsstoffe: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen (TRBA 466). Ausg. Dezember 2010, zul. erg. GMBl. Nr. 15-20 vom 25. April 2012, S. 380.
- Chapin, A.; Rule, A.; Gibson, K.; Buckley, T.; Schwab, K.: Airborne multidrug-resistant bacteria isolated from a concentrated swine feeding operation. Environ. Health Perspect. 113 (2005) Nr. 2, S. 137-142.
- Kim, H. B.; Isaacson, R. E.: The pig gut microbial diversity: Understanding the pig gut microbial ecology through the next generation high throughput sequencing. Vet. Microbiol. 177 (2015), S. 242-251.
- Nehmé, B.; Forster, R. J.; Létourneau, V.; Veillette, M.; Duchaine, C.: Culture-independent approach of the bacterial bioaerosol diversity in the standard swine confinement buildings, and assessment of the seasonal effect. Environm. Microbiol. 10 (2008) Nr. 3, S. 665-675.
- Marti, R.; Dabert, P.; Pourcher A.-M.: Pig manure contamination marker selection based on the influence of biological treatment on the dominant fecal microbial groups. Appl. Environm. Microbiol. 75 (2009) Nr. 15, S. 4967-4974.
Dr. Andrea Gärtner, Dipl.-Phys. Ing. Andreas Gessner, B. Eng. Sandra Knust - Landesamt für Natur, Umwelt und Verbraucherschutz Nordrhein-Westfalen, Dienstort Essen.
PD Dr. habil. Udo Jäckel, M. Sc. Silvana Gromöller, Dipl.-Biol. Dipl.-Ing. Kerstin Klug - Bundesanstalt für Arbeitsschutz und Arbeitsmedizin, Berlin.