Bioaerosoluntersuchungen aus der Emission einer Putenmastanlage
Zusammenfassung Die bakterielle Zusammensetzung der Bioaerosolemissionen einer Putenmastanlage mit 1 700 Tierplätzen wurde intensiv untersucht. Dabei wurden Bakterien aus den Emissionsproben von verschiedenen Nährmedien isoliert und mittels MALDI-TOF MS identifiziert. Die Sammelflüssigkeiten der Emissionsimpinger wurden außerdem einer molekularbiologischen 16S-rRNA-Gen-Analyse unterzogen. Dafür wurden über den Mastverlauf vier unabhängige Klonbibliotheken erzeugt. Die kultivierten Bakterien spiegeln vorwiegend die hautassoziierte Bakteriengemeinschaft der Puten wider. Hierbei wurden vorrangig Vertreter der Gattungen Staphylococcus/Jeotgalicoccus sowie Corynebacterium stationis detektiert. In den Klonbibliotheken herrschten Sequenzen vor, die die höchste Ähnlichkeit zu Bakterienarten – der mikroaerophilen und obligat anaeroben Bakteriengemeinschaft – des Putendarms besitzen. Die Sequenzauswertung zeigte aber auch, dass Spezies der Gattungen Lactobacillus, Staphylococcus, Romboutsia, Streptococcus, Aerococcus, Megamonas, Jeotgalicoccus, Blautia, Faecalibacterium, Ruminococcus und Clostridium scheinbar zu allen Zeitpunkten der Mast emittiert werden. Ein großer Teil der Sequenzen konnte keiner bislang kultivierten Bakterienart zugeordnet werden und demonstriert damit eine bestehende Wissenslücke. Aerococcus viridans verdient für zukünftige gesundheitliche Risikobeurteilungen besondere Aufmerksamkeit. Die Implementierung potenziell neuer Leitparameter für zukünftige Messplanungen ist Gegenstand der Diskussion.

Quelle: PantherMedia/Branex
1 Einführung
Qualitative Untersuchungen von Bioaerosolen in und aus landwirtschaftlichen Betrieben können der Expositionsabschätzung von Beschäftigten sowie Anwohnern dienen. Gegebenenfalls liefern die Ergebnisse eine erste Grundlage zur Bewertung von Risiken für die menschliche Gesundheit. Das Regelwerk des VDI hat anlagenbezogene, umweltmedizinisch relevante Messparameter festgelegt, die bei Emissions- und Immissionsmessungen berücksichtigt werden sollten. Für Arbeitsplatzmessungen werden solche speziellen Messparameter nicht festgelegt. Die Auswahl dieser vom VDI festgelegten Parameter beruht auf der Wissensgrundlage aus Tierhygiene, Epidemiologie und zum Teil auf Ergebnissen von Bioaerosoluntersuchungen. Für Anlagen der Tierhaltung werden die Gruppen Staphylokokken, intestinale Enterokokken, Enterobacteriaceae und als Spezies Staphylococcus aureus als Leitparameter mit Indikatorpotenzial im Bioaerosol festgelegt [1]. Der Begriff Staphylokokken wird im Sinne des VDI-Regelwerks als Synonym für Vertreter der Gattung Staphylococcus verwendet. Der Begriff intestinale Enterokokken ist dem Fäkalindikator der Wasseranalytik angeglichen [2]. Der Gehalt an Staphylokokken korreliert dabei eng mit der Belegungsdichte und der Aktivität des Geflügels, während intestinale Enterokokken und Enterobacteriaceae einen Zusammenhang zur fäkalen Herkunft der Bakterien im Bioaerosol assoziieren.
Eine konkrete Aussage zu einem humanpathogenen Potenzial lassen die auf Summenparametern basierenden Messwerte allerdings nicht zu. Das liegt darin begründet, dass sich unter den Zielorganismen Infektionserreger und auch solche ohne bekanntes Infektionspotenzial befinden. Daher ist zunächst eine Differenzierung auf Speziesebene wichtig [3]. Somit ist lediglich Staphylococcus aureus aufgrund seiner gewebeinvasiven Aktivität und des Auftretens „methicillinresistenter“ Stämme als potenziell pathogener Leitparameter anzusehen.
Über die detaillierte Zusammensetzung der Bakteriengemeinschaften war vor Erarbeitung der VDI-Richtlinien noch sehr wenig bekannt. In den vergangenen fünf Jahren wurden in Deutschland daher intensivere mikrobiologische Untersuchungen zu den Bakteriengemeinschaften in den Abluftkaminen von Hühner- und Schweinemastställen durchgeführt [4; 5]. Für Bioaerosole aus der Putenmast fehlten bislang jedoch aussagekräftige Daten. Zum Schließen u. a. dieser Wissenslücke wurde deshalb zunächst ein zweijähriges Forschungsprojekt des Landesamts für Umwelt, Landwirtschaft und Geologie (LfULG) des Freistaates Sachsen durchgeführt. Bioaerosole aus der Emission einer Putenmastanlage mit 1 700 Tierplätzen wurden über fünf komplette Mastperioden näher analysiert. Die quantitativen Ergebnisse wurden bereits in der Schriftenreihe des LfULG „Bioaerosole aus Anlagen der Geflügelhaltung“ [6] veröffentlicht. Mikrobiologische Kennwerte sowie Emissionsfaktoren aus dieser Anlage wurden von Lippmann und Mietke publiziert [7]. Die ermittelten Emissionsfaktoren aus diesen Untersuchungen gingen in die Richtlinie VDI 4255 Blatt 3 [8] für zwangsbelüftete Putenhaltungsanlagen ein. Im Jahresdurchschnitt lagen die Mediane für die geforderten Leitparameter bei 8,3 x 105 KBE/m³ Staphylokokken, 6,4 x 104 KBE/m³ Enterokokken, 4,0 x 102 Enterobacteriaceae/m³ und 7,4 x 101 Staphylococcus aureus/m³ (KBE: Kolonie bildende Einheiten).
Mit dem hier vorgestellten zweiten Teil der Studie erfolgt die weitergehende Charakterisierung der Bakterien im Putenstall-Bioaerosol. Erste kulturabhängige und molekularbiologische Untersuchungen wurden bereits von Fallschissel et al. [9] durchgeführt. Hierbei wurden sehr komplex zusammengesetzte Bakteriengemische identifiziert. In den generierten 16S-rRNA-Gen-Klonbibliotheken wurden überwiegend Sequenzen mit höchster Ähnlichkeit zu Vertreten der Gattungen Lactobacillus, Aerococcus, Megamonas, Staphylococcus und Weissella detektiert. Um einen weitergehenden Einblick zu erzielen, wurden in der hier vorliegenden Arbeit die Bakterienisolate, die in der beschriebenen sächsischen Studie von den in den VDI-Richtlinien empfohlenen Nährmedien gewonnen wurden, näher identifiziert. Parallel wurden Sammelflüssigkeiten der eingesetzten Emissionsimpinger einer molekularbiologischen 16S-rRNA-Analyse unterzogen.
2 Material und Methoden
Umfassende Angaben zu technischen Daten des Mastputenstalls, der Durchführung der Messungen sowie der mikrobiologischen Analytik sind in [7] beschrieben.
2.1 Isolierung und Identifizierung der Bakterien
Zur Ermittlung der Gesamtbakterienzahl sowie der Leitparameter Staphylokokken und Enterokokken erfolgte die Kultivierung gemäß den Vorgaben der Richtlinie VDI 4253 Blatt 3 [10] auf Casein-Sojapepton-Agar, Mannit-Kochsalz-Agar und Slanez-Bartley-Agar im Oberflächenverfahren. Von den gewachsenen Kolonien dieser Nährmedien wurden Subkulturen auf Columbia-Agar mit Schafblut bei 37 °C angelegt und auf Reinheit überprüft. Die Differenzierung dieser Subkulturen erfolgte mittels MALDI-TOF MS (VITEK MS Plus, bioMèrieux) durch die RIPAC-LABOR GmbH. Isolate, die nicht in der dortigen SARAMIS-Datenbank enthalten waren, wurden mittels 16S Sequenzierung identifiziert.
Der Leitparameter Enterobacteriaceae wurde gemäß der Richtline VDI 4253 Blatt 3 [10] analysiert. In Anlehnung an DIN EN ISO 21528-1 [11] wurde hierfür außerdem eine Anreicherung in gepuffertem Peptonwasser im MPN-Verfahren vorgenommen. Die beimpften Röhrchen wurden bei 37 °C über 24 h bebrütet. Anschließend erfolgte ein Ausstrich aus der Anreicherung jedes beimpften Röhrchens auf Mc-Conkey- Agar Nr. 3. Der beimpfte Nährboden wurde bei 37 °C über weitere 24 h kultiviert. Gewachsene Kolonien wurden auf Nähragar 1 subkultiviert und durch Oxidasereaktion und Glucosevergärung bestätigt.
Der Leitparameter S. aureus wurde gemäß der Vorgabe in [10] im Oberflächenverfahren auf Baird-Parker- Agar analysiert. Außerdem wurde in Anlehnung an das vom Bundesinstitut für Risikobewertung (BfR) empfohlene zweistufige Anreicherungsverfahren zum Nachweis von Methicillin-resistenten S. aureus (MRSA) [12] ein erster Anreicherungsschritt in Mueller-Hinton-Bouillon mit 6,5% NaCl im MPN-Verfahren durchgeführt. Die mit Bioaerosolflüssigkeit aus drei Verdünnungsstufen beimpften Röhrchen wurden bei 37 °C über 24 Stunden bebrütet. Anschließend erfolgte ein Ausstrich aus der Anreicherung auf Baird-Parker-Agar und Baird-Parker-Agar mit RPF (Rabbit Plasma Fibrinogen) und Kultivierung bei 37 °C über weitere 24 Stunden. Verdächtige Kolonien wurden dem Koagulasetest unterzogen.
2.2 Erstellung der Klonbibliotheken
Von Bioaerosolproben aus der 7., 10., 14. und 19. Lebenswoche der Puten (zweite Mastperiode) wurden jeweils unabhängige 16S-rRNA-Genbibliotheken mit je 300 Klonen erstellt. Die Probenaufarbeitung folgte dabei dem in Martin et. al. [13] beschriebenem Vorgehen. Die molekularbiologische Analyse von Bioaerosolen ist in [14 bis 16] hinreichend beschrieben. Die Klonierungsschritte und Sequenzierungsreaktionen erfolgten durch das Auftragslabor LGC-Genomics, Berlin.
3 Zusammensetzung der Bakteriengemeinschaft in den Bioaerosolproben aus der Putenmast
3.1 Kultivierte Bakterien in Abhängigkeit vom Nährmedium
3.1.1 Casein-Sojapepton-Agar (Gesamtbakterien)
Aus den Untersuchungen in der 19. Lebenswoche der fünften Mastperiode wurden alle gewachsenen Kolonien beider Nährbodenplatten der Verdünnungsstufe 10-3 subkultiviert und differenziert (n = 116). Der Gesamtbakteriengehalt dieser Probe lag bei 5,9 x 107 KBE/m3 Luft. Tabelle 1 gibt die identifizierten Spezies in der Reihenfolge ihres Auftretens – unabhängig von der Gattungszugehörigkeit – wieder.
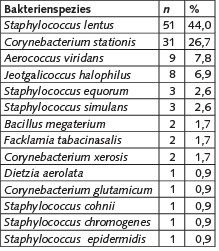
Tabelle 1. Identifizierte Bakterienspezies sowie deren Häufigkeit und relativer Anteil aller gewachsener Kolonien auf Casein-Sojapepton-Agar. Die untersuchten Emissionsproben wurden in der 19. Lebenswoche der Puten der fünften Mastperiode entnommen. Die Kultivierung erfolgte bei 37 °C für einen Zeitraum von 48 h. Die Isolierung erfolgte auf Columbia-Agar mit Schafblut bei 37 °C.
3.1.2 Mannit-Kochsalz-Agar (Staphylokokken)
Die Subkulturen stammen aus der 19. Lebenswoche der vierten Mastperiode sowie der 7., 12. und 19. Lebenswoche der fünften Mastperiode. Erfasst wurden jeweils alle Kolonien beider Nährbodenplatten der Verdünnungsstufe 10-3. Der Anteil der identifizierten Staphylokokken an allen gewachsenen Kolonien variierte in Abhängigkeit vom Lebensalter der Tiere zwischen 30,6 und 94,5 %. S. lentus, S. cohnii und S. equorum wurden in allen Proben bzw. zu allen Zeitpunkten detektiert, während S. xylosus, S. saprophyticus und S. arlettae jeweils nur zu zwei Zeitpunkten der Mast erfasst wurden. S. epidermidis, S. chromogenes, S. hominis und S. carnosus wurde jeweils nur in einer Probe detektiert (Tabelle 2).
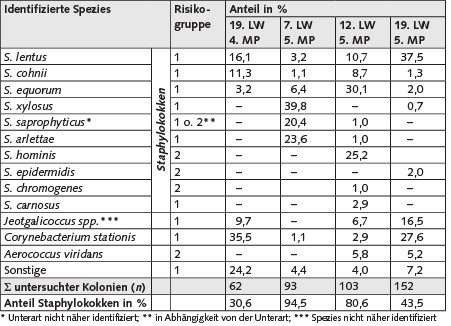
Tabelle 2. Identifizierte Bakterienspezies sowie deren relativer Anteil an allen auf Mannit-Kochsalz-Agar gewachsenen Kolonien, die Summe der untersuchten Kolonien und relativer Anteil der Staphylokokken. Die untersuchten Emissionsproben wurden in der 19. Lebenswoche der Puten der vierten Mastperiode (MP) sowie der 7., 12. und 19. Lebenswoche (LW) der fünften Mastperiode entnommen. Die Kultivierung erfolgte bei 37 °C für einen Zeitraum von 48 h. Die Isolierung erfolgte auf Columbia-Agar mit Schafblut bei 37 °C.
3.1.3 Slanez-Bartley-Agar (Enterokokken)
Der Enterokokken-Nährboden nach Slanez und Bartley ist ein Selektivmedium zum Nachweis und zur Zählung von Enterokokken, das in der Richtlinie VDI 4253 Blatt 3 [10] zur Quantifizierung des Leitparameters Enterokokken vorgesehen war. Gezählt werden dabei alle roten, rotbraunen und rosa Kolonien, die nach 48 h bei 37 °C gewachsen sind und sich als grampositive, katalase-negative Kokken erweisen. Aus der Untersuchung in der 19. Lebenswoche der fünften Mastperiode wurden alle Kolonien dieses Morphotypen von beiden parallel beimpften Nährbodenplatten der ersten Verdünnungsstufe durch Subkultivierung isoliert und identifiziert (n = 47). Davon erwiesen sich 24 Isolate (51%) als Enterococcus hirae und 23 Kolonien (49 %) als Aerococcus viridans.
3.1.4 Enterobacteriaceae
Enterobacteriaceae konnten auf Mc-Conkey-Agar im Oberflächenverfahren generell nicht nachgewiesen werden. Nach Anreicherung im MPN-Verfahren gelang eine semiquantitative Aussage. Die Konzentrationen bewegten sich zwischen 1,5 x 101 und 3,0 x 104/ m³ Luft. Es konnte weder ein jahreszeitlicher noch ein altersabhängiger Zusammenhang festgestellt werden. Die Variationen ergaben sich aus Aktivitäten des Stallmanagements (Entmistung, Erneuerung der Einstreu) [6]. Eine Speziesdifferenzierung dieses Leitparameters erfolgte nicht.
3.1.5 Staphylococcus aureus
Der direkte Nachweis von S. aureus im Oberflächenverfahren gelang aufgrund der massiven Begleitflora nicht. Die ermittelten Konzentrationen im semiquantitativen MPN-Verfahren variierten zwischen 8,1 x 100 und 1,1 x 105/m³ Luft. Dabei konnte der Leitparameter bis zur zehnten Lebenswoche nie isoliert werden, während die Messungen gegen Ende der Mast (bis 20. Lebenswoche) immer quantifizierbare Ergebnisse erbrachten [6].
3.2 Klonbibliotheken
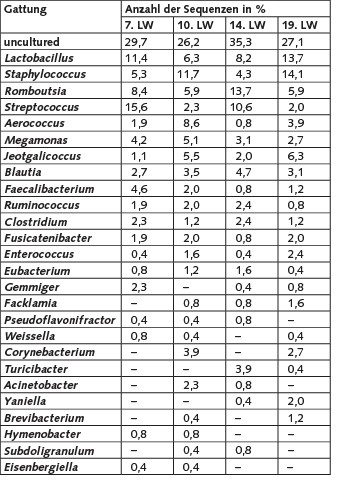
Insgesamt wurden die etwa 1 200, aus vier Proben analysierten Sequenzen 48 verschiedenen Gattungen zugeordnet. Zu den Gattungen, die in mindestens zwei der vier Klonbibliotheken erfasst wurden, zählen Lactobacillus, Staphylococcus, Romboutsia, Streptococcus, Aerococcus, Megamonas, Jeotgalicoccus, Blautia, Faecalibacterium, Ruminococcus, Clostridium, Fusicatenibacter, Enterococcus, Eubacterium, Gemmiger, Facklamia, Pseudoflavonifractor, Weissella. Corynebacterium, Turicibacter, Acinetobacter, Yaniella, Brevibacterium, Hymenobacter, Subdoligranulum und Eisenbergiella. Die angegebene Reihenfolge spiegelt dabei die Häufigkeit und den relativen Anteil der Sequenzen in den Klonbibliotheken wider (Tabelle 3).
Sequenzen, die den Gattungen Kurthia, Gallicola, Intestinibacter, Atopostipes, Wautersiella, Anaerostipes, Anaerococcus, Chryseobacterium, Enteractinicoccus, Jeotgalibaca, Nosocomiicoccus, Salinicoccus, Savagea, Sporosarcina, Alcaligenes, Dietzia, Lysinibacillus, Pediococcus, Streptomyces, Citrobacter, Microbacterium und Shigella zugeordnet wurden, konnten nur zu einem Messzeitpunkt mit relativen Häufigkeit zwischen 0,4 und 2,7% detektiert werden und sind hier nicht gesondert dargestellt.
Den größten Anteil aller Sequenzen in den Klonbibliotheken nehmen jedoch solche ein, die < 95 % Sequenzähnlichkeiten zu einem bisher gültig beschriebenen Bakterium besitzen („uncultured“). Damit können sie als Vertreter unbekannter Gattungen vermutet werden. Eine nähere Abschätzung der Anzahl dieser Gattungen erfolgte im Rahmen dieser Studie allerdings nicht. Die relativen Häufigkeiten dieser Sequenzen reichen in den Klonbibliotheken von 26,2 bis 35,3 %.
Die Zusammensetzung der Bakteriengemeinschaft aus den vier Klonbibliotheken wurde weiterhin auf Artebene vorgenommen. Als „uncultured“ wurden die Sequenzen zusammengefasst, die eine Sequenzähnlichkeit < 97 % zu bisher gültig beschriebenen prokaryontischen Typstämmen aufweisen. Insgesamt wurden die Sequenzen 91 verschiedenen Spezies zugeordnet. Die 16S-rRNA-Gensequenzen, die in allen vier untersuchten Bioaerosolproben vorkommen, sind solche mit höchsten Ähnlichkeiten zu Lactobacillus salivarius, Lactobacillus aviarius subsp. aviarius, Lactobacillus crispatus, Staphylococcus lentus, Romboutsia ilealis, Streptococcus alactolyticus, Aerococcus viridans, Megamonas hypermegale, Jeotgalicoccus halophilus, Blautia glucerasea, Ruminococcus torques und Clostridium glycyrrhizinilyticum.
Den größten Anteil an Sequenzen in den Klonbibliotheken nehmen solche ein, die keiner bisher gültig beschriebenen Bakterienspezies zuzuordnen sind. Sie haben eine relative Häufigkeit von 37,6 bis 49,4 % in der Klonbibliothek.
Auf der Basis der höchsten Sequenzähnlichkeit wurden 70 der 91 Sequenzen Bakterienarten der Risikogruppe 1 und 18 Bakterienarten der Risikogruppe 2 nach TRBA 466 [17] zugeordnet. Drei Sequenzen hatten die größte Sequenzähnlichkeit zu Bakterienarten, die bislang noch keiner Risikogruppe zugeordnet wurden.
16S rRNA-Gensequenzen mit der höchsten Ähnlichkeit zu Aerococcus viridans (Risikogruppe 2) konnten in allen vier Mastabschnitten nachgewiesen werden.
Neben 16S-rRNA-Gensequenzen mit höchster Ähnlichkeit zu L. salivarius und L. aviarius subsp. aviarius wurden Sequenzen detektiert, die weiteren Spezies der Gattung Lactobacillus am ähnlichsten waren. Hierzu gehörten Sequenzen mit höchster Ähnlichkeit zu L. crispatus, L. gasseri, L. reuteri, L. agilis, L. ingluviei, L. johnsoni, L. taiwanensis, L. vaginalis, L. sakei subsp. carnosus und L. pontis.
4 Diskussion
4.1 Staphylokokken
Die in den Bioaerosolproben vorhandenen kultivierbaren Bakterien auf Mannit-Kochsalz-Agar und Casein-Sojapepton-Agar sind sehr ähnlich (Tabellen 1 und 2). So wurden ausschließlich gram-positive, halophile Bakterien detektiert. Diese sind größtenteils auf die hautassoziierte Bakteriengemeinschaft der Tiere zurückzuführen. Mit Ausnahme von S. hominis und S. carnosus wurden in verschiedenen Arbeiten bereits alle kultivierbaren Staphylokokken als Besiedler der Haut von Geflügel beschrieben [18 bis 23]. Mannit-Kochsalz- Agar mit seinem hohen Kochsalzgehalt von 7,5 % ist kein Differentialnährboden für Staphylokokken, kann aber scheinbar die vorhandene aerobe halophile Bakteriengemeinschaft sehr gut abbilden. In zahlreichen Untersuchungen in den Geflügelställen des sächsischen Forschungsprojekts lagen die Staphylokokken-Gehalte auf Mannit-Kochsalz-Agar sogar höher als der Gesamtbakteriengehalt auf Casein-Sojapepton-Agar [6]. Unabhängig von der Methode konnten als Vertreter der Gattung Staphylococcus S. lentus, S. cohnii, S. equorum, S. xylosus, S. saprophyticus, S. arlettae und S. hominis detektiert werden. Demgegenüber konnten S. epidermitis, S. chromogenes, S. carnosus und S. simulans nur über die Kultivierung und S. capitis sowie S. succinus nur über die Molekularbiologie nachgewiesen werden. Beide Methoden liefern einen sehr guten Überblick über die Vertreter der Gattung Staphylococcus und ergänzen sich.
Neben Spezies der Gattung Staphylococcus wurden aber auch Vertreter der Gattung Jeotgalicoccus sowohl kultivierungsabhängig als auch molekularbiologisch erfasst. Diese traten beim kultivierungsabhängigen Ansatz vor allem im letzten Mastabschnitt verstärkt in Erscheinung. Vertreter dieser Gattung sind grampositive katalasepositive Kokken, die gemeinsam mit Spezies der Gattungen Staphylococcus, Macrococcus, Salinicoccus und Nosocomiicoccus die Familie der Staphylococcaceae bilden. Im Routinelabor sind Vertreter der Gattung Jeotgalicoccus mit einfachen Standardtests nicht von Spezies der Gattung Staphylococcus zu unterscheiden und können somit als „falsch positive“ Staphylokokken gezählt werden [24].
Auf der Basis der molekularbiologischen Untersuchungen wurden auch Sequenzen detektiert, die den beiden weiteren beschriebenen Gattungen Salinicoccus und Nosocomiicoccus der Familie Staphylococcaceae zuzuordnen sind. Im Zuge der Kultivierung konnten solche Spezies jedoch nicht nachgewiesen werden. Vertreter aller Gattungen der Familie der Staphylococcaceae wurden auch in der Abluft der Hühnermastställe detektiert [3] und gehören damit scheinbar zum festen Bestandteil des Bioaerosols beim Geflügel [9; 25]. Daher sollte in der Zukunft der Leitparameterbegriff „Staphylokokken“ auf die Familie Staphylococcaceae erweitert werden.
Für den kultivierungsabhängigen Nachweis ist jedoch grundsätzlich zu beachten, dass auf dem hier verwendeten Nährmedium neben Vertretern der Gattungen Staphylococcus und Jeotgalicoccus auch Bakterien anderer Bakterienfamilien wachsen können. So wurden vor allem gegen Ende der Mastperiode Corynebacterium stationis und Aerococcus viridans erfasst. Bei den „sonstigen“ Isolaten (Tabelle 2) dominierte Brevibacterium epidermidis (Daten nicht gezeigt). Corynebacterium stationis, ehemals Brevibacterium stationis, ist ein grampositives Stäbchen, das in Anwesenheit von 6 bis 10 % NaCl wachsen kann [26]. Brevibacterium epidermidis wird als Vertreter der residenten Hautflora des Menschen beschrieben [27]. Es ist zu vermuten, dass diese beiden Bakterienarten die Haut der Puten besiedeln. Aerococcus viridans wird als typischer „airborne“-Mikroorganismus beschrieben [28], der in Anwesenheit von 6,5 % NaCl wächst [29], beim Geflügel eine große Rolle spielt [30; 31], aber auch in der Abluft der Schweinemast detektiert wurde [5]. Sie sind kulturmorphologisch den Staphylokokken sehr ähnlich. Daher ist es notwendig, an einer vorgegebenen Anzahl von Isolaten einer Probe Bestätigungsanalysen durchzuführen, damit eine sichere Zuordnung und Quantifizierung mit den vorgegebenen Nährmedien möglich wird. Hierfür sind bereits minimale Standards in der überarbeiteten Richtlinie VDI 4253 Blatt 3 gefordert [33].
4.2 Enterokokken
Zur Quantifizierung für die luftgetragenen Enterokokken wurde seinerzeit der Enterokokken-Nährboden nach Slanez und Bartley in der Richtlinie VDI 4253 Blatt 3 [10] vorgeschlagen. Aus der Differenzierung der Kolonien zeigte sich jedoch, dass sich 49 % der gewonnenen Isolate in dieser Untersuchung als Aerococcus viridans erwiesen. Das Bakterium gehört wie die Enterokokken zu den aerotoleranten, grampositiven, katalasenegativen Kokken, das auf vielen Nährböden gut wächst und leicht mit Enterkokken verwechselt werden kann [32]. Wie groß der anteilige Fehler bei bisherigen Bioaerosoluntersuchungen aus Tierproduktionsanlagen war, ist sicher tierart- und bestandsabhängig. In Analysen zum Aufklären dieses Phänomens konnten in Schweinemastanlagen Fehlerraten von 75 bis 100 % festgestellt werden (Daten hier nicht gezeigt). Mit der Neufassung der Richtlinie VDI 4250 Blatt 3 [1] wurde der bisherige Leitparameter „Enterokokken“ in „intestinale Enterokokken“ umbenannt. In Anlehnung an die Trinkwasserhygiene werden darunter all jene Enterokokken verstanden, die nach Subkultivierung von Slanez-Bartley-Agar auf Galle-Äsculin-Azid-Agar bei 44 °C nach zwei Stunden eine Schwarzfärbung aufweisen. Enterococcus hirae, der ebenfalls aus den Bioaerosolen der hier vorliegenden Studie auf diesem Nährmedium kultiviert wurde, gehört in die Gruppe der intestinalen Enterokokken [2]. So sollte sich die beschriebene Differenzierung auch in der Bioaerosolanalytik als sinnvoll erweisen. Diese Methodik wurde daher bei der Überarbeitung der Richtlinie VDI 4253 Blatt 3 [33] bereits berücksichtigt.
4.3 Enterobacteriaceae
Sequenzen mit höchster Ähnlichkeit zu Vertretern der Enterobacteriaceae wurden in den angelegten Klonbibliotheken nur vereinzelt gefunden. Durch die Anreicherung im MPN-Verfahren lag die Konzentration von Enterobacteriaceae im Jahresmittel in der betrachteten Putenmastanlage lediglich bei 4 x 102 vermehrungsfähigen Einheiten/m³ Luft [6]. Diese übereinstimmenden niedrigen Ergebnisse verdeutlichen, dass Enterobacteriaceae keinen geeigneten Leitparameter für Bioaerosole aus Tierhaltungsanlagen darstellt. Bei den häufigsten Gattungen in den Klonbibliotheken erwiesen sich lediglich Megamonas, Gemmiger, Acinetobacter und Hymenobacter als gramnegativ. Inwieweit diese Darmbewohner die hohen Endotoxinkonzentrationen verursachen können, die in der Luft mit einem Jahresmittel von 2,2 x 103 EU/m³ Luft gemessen wurden [6], bleibt Spekulation.
4.4 Staphylococcus aureus
Der Leitparameter Staphylococcus aureus konnte weder kulturell noch molekularbiologisch mit den beschriebenen Methoden nachgewiesen werden. Der Erreger spielt somit quantitativ in der betrachteten Putenmastanlage vermutlich keine bedeutende Rolle. Sein Anteil lag im Durchschnitt bei einem Tausendstel bis Zehntausendstel bezogen auf die kultivierbare Gesamtstaphylokokkenpopulation und konnte daher nur über eine Spezialanreicherung im MPN-Verfahren semiquantitativ dargestellt werden. Im Jahresmittel lag die Konzentration von S. aureus in der Putenmastanlage bei 74 vermehrungsfähigen Einheiten/m³ Luft [6]. S. aureus ist einer von vielen Staphylokokken, die in die Risikogruppe 2 eingruppiert sind. Seine Rolle als Leitparameter erhielt die Spezies dadurch, dass einige Vertreter Resistenzen gegenüber β-Lactam-Antibiotika (MRSA) entwickelt haben und deshalb im besonderen Fokus der Medizin stehen. Für eine fundierte Risikoabschätzung des Bioaerosols genügt allerdings der Nachweis oder das Fehlen von S. aureus nicht.
4.5 Bakteriengemeinschaft und mögliche Leitparameter für die Putenmast
Die Zusammensetzung der Bakteriengemeinschaft aus den phylogenetischen Untersuchungen der Klonbibliotheken vermittelt insgesamt noch einen weiteren Einblick in die Bakteriengemeinschaft. Wenngleich die Reihenfolge der am häufigsten ermittelten Gattungen und Spezies aufgrund spezieller analytischer Besonderheiten in der Molekularbiologie nicht unbedingt das Verhältnis in der Originalprobe widerspiegelt [13], so lässt sich feststellen, dass das Bioaerosol der Putenmast eindeutig durch Bakterien des Darmtrakts der Tiere bestimmt wird. So wurden vor allem die im Bioaerosol detektierten Bakteriengattungen Lactobacillus, Streptococcus, Megamonas, Blautia, Faecalibacterium, Ruminococcus, Subdoligranulum, Enterococcus, Eubacterium, Acinetobacter und Clostridium auch im Intestinaltrakt der Pute nachgewiesen [34 bis 36]. Dabei handelt es sich zum Teil um aerotolerante, zumeist aber um Vertreter mit obligat anaerobem Stoffwechsel, die aufgrund der gewählten Kultivierungsbedingungen nicht nachgewiesen werden konnten. Die große Sequenzgruppe „uncultured“ besteht vermutlich ebenfalls aus Prokaryonten des Darmtrakts, deren Kultivierung und Differenzierung bisher noch nicht gelang. Diese Tatsache erklärt u. a. auch die großen Differenzen zwischen den ermittelten Gesamtzellzahlen und der auf Casein-Sojapepton-Agar kultivierbaren Gesamtbakterienzahl. Während der Projektlaufzeit wurden insgesamt neun Messungen der Gesamtzellzahl mittels DAPI-Färbung gemäß Richtlinie VDI 4253 Blatt 4 [37] aus der Putenmastanlage vorgenommen. Der berechnete Median dieser Messungen lag bei 5,0 x 107 Zellen pro m³ und damit im Mittel um das 27-Fache über der kultivierungsabhängig erfassten Gesamtbakterienzahl in den gleichen Proben [6]. Insgesamt zeigen die Ergebnisse eine sehr gute Übereinstimmung mit der bereits in der Einleitung erwähnten Studie von Fallschissel et al. [9].
4.6 Lactobacillus
Besonders hilfreich für die Frage nach zukünftigen Leitparametern für Putenmastanlagen ist der hohe Anteil an 16S-rRNA-Gensequenzen mit höchster Ähnlichkeit zu Vertretern der Gattung Lactobacillus. Die wichtigsten Vertreter sind hierbei wohl Lactobacillus salivarius, Lactobacillus aviarius subsp. aviarius und Lactobacillus crispatus, da die Sequenzen mit der größten Ähnlichkeit zu diesen drei Arten in allen Proben nachgewiesen wurden (Daten im Detail nicht gezeigt). Ihre Anwesenheit ist sehr plausibel, da in Untersuchungen des Intestinaltraktes von Geflügel ebenfalls L. salivarius und L. aviarius detektiert wurden [34; 38; 39]. Vertreter der Gattung Lactobacillus sind bis auf wenige Ausnahmen aerotolerant und lassen sich gut kultivieren. So zeigen erste Untersuchungen von Bioaerosolen in Schweinemastanlagen die kulturelle Nachweisbarkeit von Spezies der Gattung Lactobacillus auf MRS-Agar (pH 5,7) bei 37 °C unter sauerstoffreduzierten Bedingungen (O2-Gehalt 6,2 bis 13,2 %) (Daten nicht gezeigt).
4.7 Clostridium
Auch Vertreter der Gattung Clostridium gehören scheinbar zu den dominierenden Bakterien von Bioaerosolen aus Putenmastanlagen. So konnten 16S-rRNA-Gensequenzen mit höchster Ähnlichkeit zu dieser Gattung mit einem Anteil zwischen 1,2 und 2,4 % in allen Klonbibliotheken nachgewiesen werden. Ihre Eigenschaft der Sporenbildung macht sie in der aeroben Umwelt vermutlich sehr resistent. Ihr kultureller Nachweis im Bioaerosol aus Schweinemastanlagen ist in ersten Untersuchungen ebenfalls gelungen (Daten nicht gezeigt). Da diese Gattung zahlreiche pathogene Spezies beinhaltet, wäre ihr Nachweis aus Sicht der gesundheitlichen Bewertung von Bioaerosolen aus Tierproduktionsanlagen zukünftig sicher hilfreich.
4.8 Aerococcus viridans
Der stete molekularbiologische Nachweis in allen Mastabschnitten sowie der kulturelle Nachweis „als Begleitorganismus“ auf vielen unterschiedlichen Nährmedien hebt Aerococcus viridans als Bakterium der Risikogruppe 2 als besonderen Leitorganismus heraus. In der Zukunft sollte dieser bei Geflügelanlagen gesondert erfasst und zur Risikoabschätzung herangezogen werden. Beim Menschen wird der Erreger im Zusammenhang mit infektiöser Endokarditis sowie Infektionen des Urogenitaltrakts erwähnt. Im klinischen Routinelabor wurde der Erreger bisher häufig als Streptococcus oder Enterococcus spp. aus den bereits genannten Gründen falsch diagnostiziert [32; 40]. Erst der Einsatz der MALDI-TOF MS-Technik führte zu einer richtigen Erkennung des Erregers. Seitdem steigt die Bedeutung der Gattung Aerococcus als humanpathogene Bakterien [40]. Die Bioaerosolforschung steht hier zukünftig vor einer neuen Herausforderung.
5 Resümee
Emissionsuntersuchungen aus Tierhaltungsanlagen sind, abgesehen von sämtlichen Klimafaktoren, besonders von der Aktivität der Tiere, deren Lebensalter und Gesundheitszustand, Frischegrad und mikrobiologischer Beschaffenheit vorhandener Einstreu, Zeitpunkt der letzten Entmistung, Fütterungsregime und anderen prozessbedingten Einflüssen abhängig. Daher stellen Bioaerosoluntersuchungen nach kurzer Probenahme, wie sie durch die Richtlinie VDI 4257 Blatt 2 [41] beschrieben ist, aus quantitativer und qualitativer Sicht immer Momentaufnahmen der jeweils vorherrschenden Situationen dar. Dennoch zeichnen sich aus den hier vorliegenden Untersuchungsergebnissen und dem Vergleich zur Studie von Fallschissel et al. [9] bestimmte bakterielle Gattungen und Arten für die Putenmast ab, die bei kulturellen und molekularbiologischen Untersuchungen in allen Mastdurchgängen erfasst wurden und als Konstante bezeichnet werden können. Zu den bislang nicht weiter berücksichtigten Gattungen, die als Leitparameter evtl. für Putenmastställe geeignet sind, zählen vor allem die Gattungen Lactobacillus und Clostridium sowie die einzelne Spezies Aerococcus viridans. Für die Zukunft gilt es jedoch, die Spezifität und Selektivität der Nährmedien sowie die Weiterentwicklung der molekularbiologischen Methoden für den quantitativen Nachweis dieser Organismen für unterschiedliche Bioaerosole zu optimieren.
Danksagung
Unser Dank gilt Frau Kerstin Klug und Silvana Gromöller für die exzellente Unterstützung bei den molekularbiologischen Analysen.
Literatur
- VDI 4250 Blatt 3: Bioaerosole und biologische Agenzien – Anlagenbezogene, umweltmedizinisch relevante Messparameter und Beurteilungswerte. Berlin: Beuth 2016.
- DIN EN ISO 7899-2: Wasserbeschaffenheit, Nachweis und Zählung von intestinalen Enterokokken – Teil 2: Verfahren durch Membranfiltration. Berlin: Beuth 2000.
- Schneider, D.; Jäckel, U.; Gärtner, A.; Dieterich, F.: Taxonomische Charakterisierung luftgetragener Bakterien der Familie Staphylococcaceae in Emissionen von Hähnchenmastanlagen. Gefahrstoffe – Reinhalt. Luft 75 (2015) Nr. 9, S. 340-346.
- Gärtner, A.; Gessner, A.; Martin, E.; Schneider, D.; Jäckel, U.: Emissionen aus der Hähnchenmast – Untersuchung zur Zusammensetzung der Bakteriengemeinschaft und Antibiotikaresistenz – Teil 2: Ergebnisse. Gefahrstoffe – Reinhalt. Luft 74 (2014) Nr. 9, S. 377-383.
- Gärtner, A.; Gessner, A.; Gromöller, S.; Klug, K.; Knust, S.; Jäckel, U.: Emissionen aus Schweinemastanlagen – Untersuchungen zur Zusammensetzung der Bakteriengemeinschaft und Antibiotikaresistenz. Gefahrstoffe – Reinhalt. Luft 76 (2016) Nr. 1/2, S. 31-38.
- Lippmann, J.; Mietke-Hofmann, H.; Deichmann, J.: Bioaerosole aus Anlagen der Geflügelhaltung. Schriftenreihe des LfULG, Heft 13/2016. https://publikationen.sachsen.de/bdb/artikel/26748
- Lippmann, J.; Mietke-Hofmann, H.: Bioaerosoluntersuchungen aus der Emission einer Putenmastanlage. Teil 1: Mikrobiologische Kennwerte und Emissionsfaktoren. Gefahrstoffe – Reinhalt. Luft 76 (2016) Nr. 9, S. 359-364.
- VDI 4255 Blatt 3: Bioaerosole und biologische Agenzien – Emissionsfaktoren für Geflügelhaltung. Berlin: Beuth 2016.
- Fallschissel, K.; Klug, K.; Kämpfer, P.; Jäckel, U.: Detection of airborne bacteria in a German turkey house by cultivation- based and molecular methods. Ann. Occup. Hyg. 54 (2010), S. 1-10.
- VDI 4253 Blatt 3: Erfassen luftgetragener Mikroorganismen und Viren in der Außenluft – Verfahren zum quantitativen kulturellen Nachweis von Bakterien in der Luft – Verfahren nach Abscheidung in Flüssigkeiten. Berlin: Beuth 2008.
- DIN EN ISO 21528-1: Mikrobiologie der Lebensmittelkette – Horizontales Verfahren für den Nachweis und die Zählung von Enterobacteriaceae – Teil 1: Nachweis von Enterobacteriaceae. Berlin: Beuth 2017.
- EURL-AMR: Technical specifications on harmonized monitoring and reporting of MRSA. EFSA-Journal 19 (2012), S. 2897.
- Martin, E.; Gessner, A.; Gärtner, A.; Jäckel, U.: Molekularbiologische Charakterisierung luftgetragener Bakterien in Emissionsproben aus Hähnchenmastanlagen. Gefahrstoffe – Reinhalt. Luft 72 (2012) Nr. 3, S. 99-103.
- Jäckel, U.; Schäfer, J.; Martin, E.: Mikrobiologische Methoden zur Gewinnung von Expositionsdaten bei berufsbedingtem Kontakt mit biologischen Arbeitsstoffen an hoch belasteten Arbeitsplätzen – Teil 1: Methodenauswahl. Gefahrstoffe – Reinhalt. Luft 73 (2013) Nr. 9; S. 358-360.
- Martin, E.; Schäfer, J.; Jäckel, U.: Mikrobiologische Methoden zur Gewinnung von Expositionsdaten bei berufsbedingtem Kontakt mit biologischen Arbeitsstoffen an hoch belasteten Arbeitsplätzen – Teil 2: Qualitative Analyse von Mikroorganismen in komplexen Bioaerosolen (Genbibliotheken). Gefahrstoffe – Reinhalt. Luft 73 (2013) Nr. 9, S. 361-366.
- Schäfer, J.; Fallschissel, K.; Martin, E.; Jäckel, U.: Mikrobiologische Methoden zur Gewinnung von Expositionsdaten bei berufsbedingtem Kontakt mit biologischen Arbeitsstoffen an hoch belasteten Arbeitsplätzen – Teil 3: Spezies- oder gattungsspezifische Quantifizierung von Mikroorganismen in Bioaerosolen an Arbeitsplätzen. Gefahrstoffe – Reinhalt. Luft 73 (2013) Nr. 10, S. 423-428.
- Technische Regeln für Biologische Arbeitsstoffe (TRBA) 466: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen. GMBI. (2015) Nr. 46-50, S. 910.
- Becker, K.; Heilmann; C.; Peters, G.: Coagulase-Negative staphylococci. Clin. Microbiol. Rev. 27 (2014), S. 870-926.
- Devriese, L. A.; Hájek, V.; Oeding, P.; Meyer, S. A.; Schleifer, K. H.: Staphylococcus hyicucus (Sopolinsky 1953) comb. nov. and Staphylococcus hyicus subsp. chromogenes subsp. nov. Int. J. System. Bacteriol. 28 (1978), S. 482-490.
- Hájek, V.; Devriese, L. A.; Mordarski, M.; Goodfellow, M.; Pulverer, G.; Varaldo, P. E.: Elevation of Staphylococcus hyicus subsp. chromogenes (Devriese et al. 1978) to species status: Staphylococcus chromogenes (Devriese et al. 1978) comb. nov. Syst. Appl. Microbiol. 8 (1986), S. 169-173.
- Schleifer, K. H.; Geyer, U.; Kilpper-Bälz, R.; Devriese, L. A.: Elevation of Staphylococcus sciuri subsp. lentus (Kloos et. al.) to Species Status: Staphylococcus lentus (Kloos et al.) comb.nov.. System. Appl. Microbiol. 4 (1983), S. 382-387.
- Schleifer, K. H.; Kilpper-Bälz, R.; Devriese, L. A.: Staphylococcus arlettae sp. nov., S. equorum sp.nov. and S. kloosii sp. nov.: Three new coagulase-negative, novobiocin-resistant species from animals. System. Appl. Microbiol. 5 (1984), S. 501-509.
- Devriese, L. A.; Schleifer, K. H.; Adegoke, G. O.: Identification of coagulase-negative staphylococci from farm animals. J. Appl. Bacteriol. 58 (1985) Nr. 1, S. 45-55.
- Schwaiger, K.; Hölzel, C.; Mayer, M.; Bauer, J.: Notes on the almost unknown genus Jeotgalicoccus. Let. Appl. Microbiol. 50 (2010), S. 441-444.
- Martin, E.; Fallschissel, K.; Kämpfer, P.; Jäckel, U.: Detection of Jeotgalicoccus spp. in poultryhouse air. System. Appl. Microbiol. 33 (2010), S. 188-192.
- Bernard, K. A.; Wiebe, D.; Burdz, T.; Reimer, A.; Ng, B.; Singh, C.; Schindle, S.; Pacheco, A. L.: Assignment of Brevibacterium stationis (ZoBell and Upham 1944) Breed 1953 to the genus Corynebacterium, as Corynebacterium stationis comb.nov., and emended description of the genus Corynebacterium to include isolates that can alkalinize citrate. Int. J. Sys. Ev. Microbiol. 60 (2010), S. 874-879.
- Collins, M. D.: The Genus Brevibacterium. The Procaryotes Vol. 3. New York: Springer 2006.
- Williams, R. E. O.; Hirch, A.; Cowan, S. T.: Aerococcus, a new bacterial genus. J. Gen. Microbiol. 8 (1953), S. 475-480.
- Facklam, R.; Elliott, J. A.: Identification, classification and clinical relevance of catalase-negative, gram-positive cocci, excluding the Streptococci and Enterococci. Clin. Microbiol. Rev. 8 (1995), S. 479-495.
- Turtura, G. C.; Lorenzelli; P.: Gram-positive cocci isolated from slaughtered poultry. Microbiol. Res. 149 (1994), S. 203-213.
- Haas, D.; Posch, J.; Schmidt, S.; Wüst, G.; Sixl, W.; Feierl, G.; Marth, E.; Reinthaler, F. F.: A case study of airborne culturable microorganisms in a poultry slaughterhouse in Styria, Austria. Aerobiol. 21 (2005), S. 193-201.
- Rasmussen, M.: Aerococci and aerococcal infections. J. Inf. 66 (2013), S. 467-474.
- VDI 4253 Blatt 3 (Entwurf): Erfassen luftgetragener Mikroorganismen und Viren in der Außenluft – Verfahren zum quantitativen kulturellen Nachweis von Bakterien in der Luft – Verfahren nach Abscheidung in Flüssigkeiten. Berlin: Beuth 2017.
- Danzeisen, J. L.; Calvert, A. J.; Noll, S. L.; McComb, B.; Sherwood, J. S.; Logue, C. M.; Johnson, T. J.: Succession of the turkey gastrointestinal bacterial microbiome related to weight gain. Peer J. 1 (2013), S.1/22-22/22. DOI 10.7717/peerj.237
- Wei, S.; Morrison, M.; Yu, Z.: Bacterial census of poultry intestinal microbiome. Poultry Sci. 92 (2013), S. 671-683.
- Scupham J. A.; Patton, G.; Bent, E.; Bayles, D. O.: Comparison of the cecal microbiota of domestic and wild turkeys. Microb. Ecol. 56 (2008) Nr. 2, S. 322-331.
- VDI 4253 Blatt 4: Bioaerosole und biologische Agenzien – Bestimmung der Gesamtzellzahl mittels Fluoreszenzanalyse nach Anfärbung mit DAPI. Berlin: Beuth 2013.
- Fujisawa, T.; Shirasaka, S.; Watabe, J.; Mitsuoka, T.: Lactobacillus aviarius sp. nov.: A new species isolated from the intestine of chickens. Syst. Appl. Microbiol. 5 (1984), S. 414-420.
- Li, Y.; Raftis, E.; Canchaya, C.; Fitzgerald, G. F.; van Sinderen, D.; O’Toole, P. W.: Polyphasic analysis indicates that Lactobacillus salivarius subsp. salivarius and Lactobacillus salivarius subsp. salicinus do not merit separate subspecies status. Int. J. Syst. Ev. Microbiol. 56 (2006), S. 2397-2403.
- Mohan, B.; Zaman, K.; Anand, N.; Taneia, N.: Aerococcus viridans: A rare pathogen causing urinary tract infection. J. Clin. Diagn. Res. 11 (2017) Nr. 1. https://dx.doi.org/10.7860/JCDR/2017/23997.9229
- VDI 4257 Blatt 2: Bioaerosole und biologische Agenzien – Messen von Emissionen – Probenahme von Bioaerosolen und Abscheidung in Flüssigkeiten. Berlin: Beuth 2011.
Dr. Henriette Mietke - Staatliche Betriebsgesellschaft für Umwelt und Landwirtschaft, Nossen.
Stephanie Keck und Dr. Udo Jäckel - Bundesanstalt für Arbeitsschutz und Arbeitsmedizin, Berlin